This is an old revision of the document!
Table of Contents
Laboratorium 4: Cechy wielomianowe
Będziemy przetwarzali następujące pliki:
- xy-002 $f_{true}=-1.5x^2+3x+4$
- xy-003 $f_{true}=-1.5x^2+3x+4$
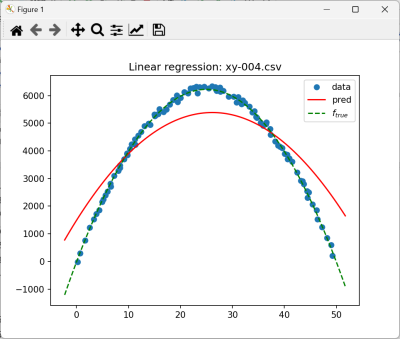
- xy-004 $f_{true}=-10x^2+500x-25$
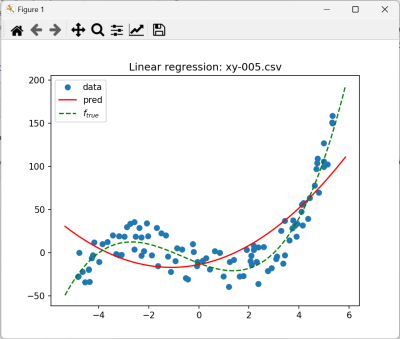
- xy-005 $f_{true}=(x+4)(x+1)(x-3)$
Wykorzystamy kod z poprzedniego laboratorium (Regresja Liniowa)
1. Cechy wielomianowe 2 stopnia
Umieść kod w klasie LinearRegressionPolynomialFeaturesOrderTwo
1. Dodaj do zbioru danych kolumnę X2 zawierającą po prostu wynik wyrażenia “X*X”
Czyli, np. dla xy-002.csv po załadowaniu i przetworzeniu danych wynikiem będzie:
+--------+----------+------------------+ | X| Y| X2| +--------+----------+------------------+ |0.411194|-59.938274| 0.169080505636| |0.549662|-72.006761| 0.302128314244| |0.860093|-68.979336| 0.739759968649| | 1.27504| 32.07157|1.6257270015999998| |2.202931|-91.531894| 4.852904990761| +--------+----------+------------------+ only showing top 5 rows root |-- X: double (nullable = true) |-- Y: double (nullable = true) |-- X2: double (nullable = true)
2. Zmień konfigurację VectorAssembler tak, aby budował wektory na podstawie kolumn [“X”, “X2”]
3. Zbuduj model regresji
4. Wyświetl wyniki. Przy wyświetlaniu aproksymowanej funkcji wywoływana jest funkcja lrModel.predict(). Jako argument musisz dostarczyć wektor zawierający zarówno wartość wejściową, jak i jej kwadrat. Możesz to zrealizować dodają parametr order do funkcji plot (przyda się przy regresji 3-stopnia) lub po prostu dodając element.
5. Przetwórz wszystkie pliki i wyświetl ich wykresy.
Niewątpliwie ciekawym wykresem jest dopasowanie wielomianu 2-stopnia do danych wygenerowanych z wielomianu 3 stopnia
ale bardziej zastanawiający jest przypadek xy-004.csv
6. Popraw dopasowanie (jest to możliwe)
Przeanalizuj parametry algorytmu. Na przykład:
LinearRegression lr = new LinearRegression() .setMaxIter(10) .setRegParam(0.3) .setElasticNetParam(0.8) .setFeaturesCol("features") .setLabelCol("Y");
Które z nich można zmienić? Sprawdź, co się stanie, gdy
- usuniemy regularyzację
- zwiększymy liczbę iteracji
- Porównaj metryki, zamieśc wykresy
Uwagi:
- usuwanie regularyzacji na ogół (dla niesyntetycznych zbiorów danych) psuje zdolność generalizacji
- zwiększenie liczby iteracji dla dużych zbiorów danych proporcjonalnie zwiększa czas uczenia
2. Cechy wielomianowe 3 stopnia
Umieść kod w klasie LinearRegressionPolynomialFeaturesOrderThree
1. Podobnie, jak w poprzednim przypadku dodaj kolumnę X3 z wartościami kolumny X podniesionymi do 3 potęgi
2. Zmodyfikuj odpowiednio VectorAssembler
3. Przeprowadź regresję (dla wszystkich zbiorów danych)
4. Zamieść wykres xy-005.csv
5. Porównaj w tabelce wartości miary r2 i MSE dla regresji 2 i 3 stopnia (dla wszystkich zbiorów danych)
- Sprawdź, czy w przypadku regresji 3-stopnia dla danych wygenerowanych z funkcji kwadratowej współczynniki przy $x^3$ były zerowe?
- Z reguły niewielkie zwiększenie współczynnika regularyzacji i liczby iteracji poprawia dopasowanie dla krzywych 2 stopnia
3.Pipeline
Kod umieść w klasie LinearRegressionPolynomialFeaturesPipeline
Ponieważ przetwarzane są kolejne zbiory, najlepiej umieść kod w funkcji postci static void processDataset(SparkSession spark,String filename,int degree,Function<Double,Double> f_true)
Dodawanie manualne cech jako kolumn zbioru danych jest zbyt żmudne - zachodzi konieczność nazywania tych kolumn, później modyfikacji kodu w kilku miejscach. Raczej buduje się ciąg przetwarzania: Pipeline, którego elementami są wstępne przetwarzanie danych i budowa modelu estymatora. Rozszerzenie cech o cechy wielomianowe jest dokonywane za pomocą odpowiedniej funkcji transformującej dane tablicowe : PolynomialExpansion.
==== Budowa ciągu przetwarzania ====
Kolejne etapy przetwarzania to:
* zastosowanie VectorAssembler
* PolynomialExpansion
* LinearRegression
Zamiast wykonywać je natychmiast utwórz i skonfiguruj odpowiednie obiekty
<code java>
VectorAssembler vectorAssembler=new VectorAssembler()
.setInputCols(new String[]{“X”})
.setOutputCol(“features”);
Dataset<Row> df_trans = vectorAssembler.transform(df);
PolynomialExpansion polyExpansion = new PolynomialExpansion()
.setInputCol(“features”)
.setOutputCol(“polyFeatures”)
.setDegree(degree);
df_trans = polyExpansion.transform(df_trans);
LinearRegression lr = new LinearRegression()
…
</code>
a następnie zdefinuj ciąg przetwarzania Pipeline i wywołaj jego metodę fit()
<code java>
Pipeline pipeline = new Pipeline()
.setStages(new PipelineStage[] {vectorAssembler, polyExpansion, lr});
PipelineModel model = pipeline.fit(df);
</code>
Aby uzyskać dostęp do konkretnego elementu ciagu - nalezy po prostu zrzutować wybrany element
<code java>
LinearRegressionModel lrModel = (LinearRegressionModel)model.stages()[2];
</code>
==== Funkcja do rysowania wykresów ====
Zmodyfikujemy sygnaturę funkcji.
<code java>
*
* @param x - współrzedne x danych
* @param y - współrzedne y danych
* @param pipelineModel - model pipeline
* @param spark - sesja Spark
* @param title - tytuł do wyswietlenia (może być null)
* @param f_true - funkcja f_true (może być null)
*/
static void plot(List<Double>x, List<Double> y, PipelineModel pipelineModel, SparkSession spark, String title, Function<Double,Double> f_true)
</code>
W odróżnieniu od funkcji używanej dotychczas - przekazujemy do niej pipelineModel. Wykorzystamy go, aby wyświetlić przebieg funkcji wyznaczonej w wyniku regresji.
1. Załóżmy, że mamy punkty, dla których mamy wyznaczyć wartości funkcji zgromadzone na liście fx
<code java>
var xdelta = 0.05*(xmax-xmin);
var fx = NumpyUtils.linspace(xmin-xdelta,xmax+xdelta,100);
</code>
Aby przetworzyć je za pomocą pipeline musimy zamienić je na Dataset<Row> zawierający kolumnę o nazwie X z odpowiednimi wartościami.
2. Zamiana wartości double na Row
Przeiteruj przez elementy fx i dla każdego z nich utwórz obiekt Row, np. wołając Row r = RowFactory.create(d); lub odpowiednią funkcje strumienia. Zbierz wynik w List<Row> rows
3. Zdefiniuj schemat i utwórz zbiór danych:
<code java>
StructType schema = new StructType().add(“X”, “double”);
Dataset<Row> df_test = spark.createDataFrame(rows,schema);
df_test.show(5);
df_test.printSchema();
</code>
Wynik:
<code>
+——————–+
| X|
+——————–+
| -2.0917333|
| -1.5355272333333332|
| -0.9793211666666666|
|-0.42311509999999974|
| 0.13309096666666687|
+——————–+
only showing top 5 rows
</code>
3. Wywołaj funkcję transform() obiektu pipeline
<code java>
Dataset<Row> df_pred = pipelineModel.transform(df_test);
df_pred.show(5);
df_pred.printSchema();
</code>
W wyniku jej wykonania do zbioru zostaną dodane kolejno kolumny features, polyfeatures i prediction zgodnie z kolejnymi etapami przetwarzania
<code>
+——————–+——————–+——————–+——————-+
| X| features| polyFeatures| prediction|
+——————–+——————–+——————–+——————-+
| -2.0917333| [-2.0917333]|[-2.0917333,4.375…| -33.40167605867543|
| -1.5355272333333332|[-1.5355272333333…|[-1.5355272333333…|-29.392038747489657|
| -0.9793211666666666|[-0.9793211666666…|[-0.9793211666666…| -26.24979833578732|
|-0.42311509999999974|[-0.4231150999999…|[-0.4231150999999…|-23.976336940609087|
| 0.13309096666666687|[0.13309096666666…|[0.13309096666666…| -22.57303667899562|
+——————–+——————–+——————–+——————-+
only showing top 5 rows
root
|– X: double (nullable = true)
|– features: vector (nullable = true)
|– polyFeatures: vector (nullable = true)
|– prediction: double (nullable = false)
</code>
4. Należy oczywiście wyświetlić zawartość kolumny prediction
===== 4. Podział na zbiór uczący i testowy =====
Podziały przetestujemy na pliku xy-003.csv dla cech wielomianowych 3 stopnia.
==== Rozwiązanie 1 ====
1. Po załadowaniu zbioru do podziel go korzystając z funkcji limit i offset:
<code java>
long rowsCount = df.count();
int trainCount = (int)(rowsCount*.7);
var df_train = df.select(“*”).limit(trainCount);
var df_test = df.select(“*”).offset(trainCount);
System.out.println(df_train.count());
System.out.println(df_test.count());
</code>
2. Zamień wywołania w kodzie tak, aby przetwarzany i wyświetlany był zbiór df_train
3. Dodaj wyświeltanie df_test:
<code java>
x = df_test.select(“X”).as(Encoders.DOUBLE()).collectAsList();
y = df_test.select(“Y”).as(Encoders.DOUBLE()).collectAsList();
plot(x,y,model,spark,String.format(“Linear regression: %s (test data)”,filename),f_true);
</code>
4. Dodaj kod odpowiedzialny za obliczanie metryk na zbiorze testowym
* wpierw przetrannsformuj go
* następnie oblicz metryki korzystając z kolumn Y i prediction
<code java>
var df_test_prediction = model.transform(df_test);
RegressionEvaluator evaluator = new RegressionEvaluator()
.setLabelCol(“Y”)
.setPredictionCol(“prediction”)
.setMetricName(“rmse”); or any other evaluation metric
double rmse = evaluator.evaluate(df_test_prediction);
evaluator.setMetricName(“r2”);
double r2 = evaluator.evaluate(df_test_prediction);
</code>
5. Wydrukuj wartości metryk i obejrzyj wykresy
<code>
Scores on training set: r2=0.63 RMSE=317.19
Scores on test set: r2=-1.02 RMSE=647.06
</code>