Table of Contents
Laboratorium 9 - klasyfikacja dokumentów tekstowych
Celem zadania jest predykcja autora tekstu na podstawie jego zawartości. Przetestujemy dwa klasyfikatory:
- Drzewo decyzyjne
- Naiwny klasyfikator Bayesa
1. Zbiory danych
Podczas ćwiczeń będziemy przetwarzali dane tekstowe pochodzące z 5 książek z przełomu XIX i XX wieku
- Reymont: Ziemia Obiecana
- Żuławski: Na srebrnym globie
- Sienkiewicz: W pustyni i w puszczy
- Sienkiewicz: Rodzina Połanieckich
- Żeromski: Syzyfowe prace
Zawartość książek została podzielona na zdania i utworzono 8 zbiorów dokumentów:
- złożonych z 10, 5, 3 i 1 zdań
- obejmujących treść wszystkich książek (five-books*.csv)
- obejmujących treść pierwszych dwóch książek (two-books*.csv)
Każdy element zbioru danych zawiera informacje o autorze, książce (work), treść (content) oraz formy podstawowe wyrazów, tzw. lematy: content_stemmed. Zbiory są zapisane w formacie UTF-8
Archiwum ZIP zawierające zbiory danych
![]() Uwaga. W przypadku pojawienia się wyjątków ( serializer kryo …) należy ustawić dodatkowe opcje VM (jak na pierwszym laboratorium). Wybieramy Edit configuration (przy przycisku Run) i dodajemy poniższe opcje w polu VM options.
Uwaga. W przypadku pojawienia się wyjątków ( serializer kryo …) należy ustawić dodatkowe opcje VM (jak na pierwszym laboratorium). Wybieramy Edit configuration (przy przycisku Run) i dodajemy poniższe opcje w polu VM options.
--add-opens=java.base/java.lang=ALL-UNNAMED --add-opens=java.base/java.lang.invoke=ALL-UNNAMED --add-opens=java.base/java.lang.reflect=ALL-UNNAMED --add-opens=java.base/java.io=ALL-UNNAMED --add-opens=java.base/java.net=ALL-UNNAMED --add-opens=java.base/java.nio=ALL-UNNAMED --add-opens=java.base/java.util=ALL-UNNAMED --add-opens=java.base/java.util.concurrent=ALL-UNNAMED --add-opens=java.base/java.util.concurrent.atomic=ALL-UNNAMED --add-opens=java.base/sun.nio.ch=ALL-UNNAMED --add-opens=java.base/sun.nio.cs=ALL-UNNAMED --add-opens=java.base/sun.security.action=ALL-UNNAMED --add-opens=java.base/sun.util.calendar=ALL-UNNAMED
na podstawie tej dyskusji
2. AuthorRecognitionDecisionTree
Przeanalizujemy zawartośc zbioru danych two-books-all-1000-10-stem.csv i zbudujemy klasyfikator oparty na algorytmie DecisionTreeClassifier.
2.1 Analiza zbioru danych
1. Załaduj zbiór danych
Dataset<Row> df = spark.read().format("csv") .option("header", "true") .option("delimiter",",") .option("quote","\'") .option("inferschema","true") .load("data/books/two-books-all-1000-10-stem.csv");
2. Wyświetl autorów i utwory (użyj funkcji select…distinct
3. Policz liczbę dokumentów poszczególnych autorów. Użyj funkcji groupBy … count. Czy zbiór jest zrównoważony?
4 Policz średnie długości tekstów w kolumnie content dla poszczególnych autorów. Dodaj wpierw kolumnę z długością, a następnie zgrupuj i zastosuj avg jako funkcję agregująca.
2.2 Tokenizacja (wydzielanie symboli)
Użyj klasy RegexTokenizer. Zwróć uwagę na separatory. W oryginalnych plikach tekstowych pojawiały się specyficzne znaki. Jeśli nie zostaną zdefiniowane jako separatory, zostaną zidentyfikowane jako słowa (lub ich części). Na ich podstawie łatwo można zidentyfikować źródło, a więc i autora.
String sep = "[\\s\\p{Punct}—…”„]+"; RegexTokenizer tokenizer = new RegexTokenizer() .setInputCol("content") .setOutputCol("words") .setPattern(sep); var df_tokenized = tokenizer.transform(df); df_tokenized.show();
2.3 Konwersja do postaci Bag of Words
Reprezentacja BoW to odwzorowanie (słowo→liczba wystąpień).
- Budowana jest za pomocą transformacji
CountVectorizer, która:- Tworzy słownik słów (zazwyczaj uporządkowany według liczby wystąpień)
- Zlicza wystąpienia danego słowa w dokumencie
- Zamienia tekst na rzadki wektor, którego i-ty element to liczba wystąpień i-tego słowa w słowniku
1. Zdefiniuj parametry i przekonwertuj zbiór danych
CountVectorizer countVectorizer = new CountVectorizer() .setInputCol("words") .setOutputCol("features") .setVocabSize(10_000) // Set the maximum size of the vocabulary .setMinDF(2); // Set the minimum number of documents in which a term must appear CountVectorizerModel countVectorizerModel = countVectorizer.fit(df_tokenized); Dataset<Row> df_bow = countVectorizerModel.transform(df_tokenized); df_bow.select("words","features").show(5);
2. Wyświetl słowa i wektor features dla pierwszego wiersza (dokumentu). Wywołanie row.get(index) zwraca odpowiedni element wiersza
3. SparseVector ma metodę indices(), która zwraca indeksy niezerowych elementów. To równocześnie indeksy słów w słowniku, który możesz pobrać za pomocą countVectorizerModel.vocabulary(). Dla pierwszego wiersza wyświetl odwzorowanie (słowo→liczba wystąpień).
i -> 13.000000 się -> 8.000000 w -> 8.000000 na -> 3.000000 z -> 4.000000 do -> 3.000000 a -> 1.000000 co -> 1.000000 za -> 1.000000 jeszcze -> 2.000000 ze -> 1.000000 ...
Tego typu słowa to tzw. stopwords. Najczęściej przy kategoryzacji tekstów są one usuwane, ale niekoniecznie w przypadku identyfikajci autorów.
2.4 Budowa klasyfikatora
1. Przed przystąpieniem do klasyfikacji należy przekonwertować nazwiska autorów do postaci etykiet numerycznych
StringIndexerModel labelModel = labelIndexer.fit(df_bow); df_bow = labelModel.transform(df_bow); df_bow.show();
2. Konfiguracja algorytmu, uczenie i predykcja
DecisionTreeClassifier dt = new DecisionTreeClassifier() .setLabelCol("label") .setFeaturesCol("features") .setImpurity("gini") // lub entropy .setMaxDepth(30); DecisionTreeClassificationModel model = dt.fit(df_bow); Dataset<Row> df_predictions = model.transform(df_bow); df_predictions.show();
2.5 Ocena klasyfikatora
Użyj odpowiednio skonfigurowanej klasy MulticlassClassificationEvaluator (porównaj kolumny label i prediction). Wyznacz metryki f1 i accuracy. Spodziewane są wartości rzędu 0.99.
2.6 Istotność cech
Istotność cech (ang. feature importance) to ocena wkład danej cechy (tu słowa) na wynik klasyfikacji. W przypadku drzew decyzyjnych i lasów random forest używane są takie miary nieczystości, jak nieczystość Gini lub entropia. Istotność cechy j jest obliczana poprzez zsumowanie poprawy nieczystości we wszystkich węzłach, w których cecha j jest używana do podziału danych, ważone liczbą próbek docierających do tego węzła.
1. Odczytaj wektor istotności cech
SparseVector fi = (SparseVector) model.featureImportances(); System.out.println(fi);
2. Dla indeksów niezrowych wartości wektora fi.indices() wypisz odpowiedni element ze słownika oraz wartośc istotności.
Jakie słowa przesądzają o wyniku klasyfikacji?
ziemi -> 0,220425 marta -> 0,173971 tom -> 0,069526 piotr -> 0,054149 ada -> 0,047203 wóz -> 0,034318 borowiecki -> 0,029524 morze -> 0,027261 i -> 0,023887 zgoła -> 0,020877 człowieku -> 0,020661 pan -> 0,020352 niebie -> 0,015931 zapewne -> 0,015552 snadź -> 0,015438 gór -> 0,015401 marty -> 0,015326 wybrzeżu -> 0,012599 tomasz -> 0,012525 ziemio -> 0,009627 patrzyłem -> 0,009584 ...
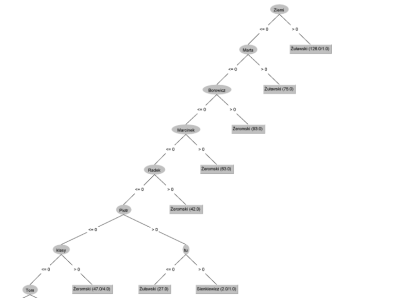
Przy użyciu Weka możliwe było wyświetlenie drzewa. Poniżej przykład dla zbioru danych five-books*.csv. Przetwarzamy two-books*.csv więc cechy są trochę inne…
3. AuthorRecognitionGridSearchCVDecisionTree
Napisz funkcję, która przeprowadzi 3-krotną walidację krzyżową połaczoną z przeszukiwaniem siatki parametrów
private static void performGridSearchCV(SparkSession spark, String filename)
1. Podziel zbiór danych w proporci 0.8 i 0.2. Na zbiorze df_train będzie przeprowadzana walidacja krzyżowa
var splits = df.randomSplit(new double[]{0.8,0.2}); var df_train=splits[0]; var df_test=splits[1];
2. Powtórz poprzednie kroki definiując odpowiednie obiekty
3. Zdefiniuj ciąg przetwarzania
Pipeline pipeline = new Pipeline() .setStages(new PipelineStage[] {tokenizer, countVectorizer,labelIndexer, decisionTreeClassifier});
4. Zdefiniuj siatkę parametrów
ParamMap[] paramGrid = new ParamGridBuilder() .addGrid(countVectorizer.vocabSize(), new int[] {100, 1000,10_000}) .addGrid(decisionTreeClassifier.maxDepth(), new int[] {10, 20,30}) .build();
5. Skonfiguruje ewaluator. Zależy nam na wysokich wartościach metryki F1
MulticlassClassificationEvaluator evaluator = new MulticlassClassificationEvaluator() .setLabelCol("label") .setPredictionCol("prediction") .setMetricName("f1");
6. Zdefiniuj parametry walidacji krzyżowej. Jej wykonanie potrwa kilka minut.
CrossValidator cv = new CrossValidator() .setEstimator(pipeline) .setEvaluator(evaluator) .setEstimatorParamMaps(paramGrid) .setNumFolds(3) // Use 3+ in practice .setParallelism(8); CrossValidatorModel cvModel = cv.fit(df);
7. Pobierz najlepszy znaleziony model i wypisz parametry jego etapów
PipelineModel bestModel = (PipelineModel) cvModel.bestModel(); for(var s:bestModel.stages()){ System.out.println(s); }
Jakie były te parametry?
- rozmiar słownika
- głębokość drzewa
Wykorzystamy je w następnym etapie.
8. Wypisz średnie wartości metryki f1 dla badanych modeli – cvModel.avgMetrics()
9. Przetestuj efektywność najlepszego model na zbiorze testowym.
Wypisz wartości:
- accuracy
- weightedPrecision
- weightedRecall
- f1
4. AuthorRecognitionCVDecisionTree
1. Zmodyfikuj kod klasy AuthorRecognitionCVGridSearch. Wpisz znalezione parametry (wielkość słownika i głębokość drzewa). Zmień nazwę funkcji performGridSearchCV na performCV
2. Siatka poszukiwań może być pusta:
CrossValidator cv = new CrossValidator() .setEstimator(pipeline) .setEvaluator(evaluator) .setEstimatorParamMaps(new ParamGridBuilder().build()) .setNumFolds(3) // Use 3+ in practice .setParallelism(8);
3. Przetestuj wydajność klasyfikatora dla wszystkich plików. Zbierz wyniki w tabelce.
String filenames[]={ "data/books/two-books-all-1000-1-stem.csv", "data/books/two-books-all-1000-3-stem.csv", "data/books/two-books-all-1000-5-stem.csv", "data/books/two-books-all-1000-10-stem.csv", "data/books/five-books-all-1000-1-stem.csv", "data/books/five-books-all-1000-3-stem.csv", "data/books/five-books-all-1000-5-stem.csv", "data/books/five-books-all-1000-10-stem.csv", };
5. NaiveBayesDemo
Przetestujemy wpierw zasadę działania klasyfikatora NB na niewielkim przykładzie
1. Utworzymy zbiór danych. Teksty aaa, bbb,… to słowa
StructType schema = new StructType() .add("author", DataTypes.StringType, false) .add("content", DataTypes.StringType, false); List<Row> rows = Arrays.asList( RowFactory.create("Ala","aaa aaa bbb ccc"), RowFactory.create("Ala","aaa bbb ddd"), RowFactory.create("Ala","aaa bbb"), RowFactory.create("Ala","aaa bbb bbb"), RowFactory.create("Ola","aaa ccc ddd"), RowFactory.create("Ola","bbb ccc ddd"), RowFactory.create("Ola","ccc ddd eee") ); var df = spark.createDataFrame(rows,schema);
2. Dalej następuje standardowy ciąg przetwarzania
String sep = "[\\s\\p{Punct}—…”„]+"; RegexTokenizer tokenizer = new RegexTokenizer() .setInputCol("content") .setOutputCol("words") .setPattern(sep); df = tokenizer.transform(df); df.show(); System.out.println("-----------"); // Convert to BoW with CountVectorizer CountVectorizer countVectorizer = new CountVectorizer() .setInputCol("words") .setOutputCol("features") .setVocabSize(10_000) // Set the maximum size of the vocabulary .setMinDF(1) // Set the minimum number of documents in which a term must appear ; // Fit the model and transform the DataFrame CountVectorizerModel countVectorizerModel = countVectorizer.fit(df); df = countVectorizerModel.transform(df); // Prepare the data: index the label column StringIndexer labelIndexer = new StringIndexer() .setInputCol("author") .setOutputCol("label"); StringIndexerModel labelModel = labelIndexer.fit(df); df = labelModel.transform(df); df.show();
3. Definicja parametrów algorytmu i uczenie
NaiveBayes nb = new NaiveBayes() .setLabelCol("label") .setFeaturesCol("features") .setModelType("multinomial") .setSmoothing(0.01) ; System.out.println(nb.explainParams()); NaiveBayesModel model = nb.fit(df);
5.1 Parametry modelu
Parametrami modelu są wartości prawdopodobieństw (logarytmów prawdopodobieństw) zwracane poprzez odpowiednie funkcje:
theta()- prawdopodobieństwo warunkowe (likelihood): $p(x_i|y_j)$pi()- prawdopodobieństwo a-priori klasy: $p(y_j)$sigma()- wartości wariancji - tylko dla rozkładu Gaussa
W przypadku rozkładu multinomial używane są tylko dwa pierwsze.
1. Wypisz informacje o zawartości słownika oraz etykietach
String[] vocab = countVectorizerModel.vocabulary(); String[] labels= labelModel.labels();
2. Następnie wpisz prawdopodobieństwa warunkowe (likelihhod) na podstawie zawartości macierzy theta. Funkcja apply() zwraca element wektora lub macierzy, np.theta.apply(i,j).
Oczekiwany wynik:
P(bbb|Ala)=0.415768 (log=-0.877629) P(aaa|Ala)=0.415768 (log=-0.877629) P(ddd|Ala)=0.083817 (log=-2.479114) P(ccc|Ala)=0.083817 (log=-2.479114) P(eee|Ala)=0.000830 (log=-7.094235) P(bbb|Ola)=0.111602 (log=-2.192814) P(aaa|Ola)=0.111602 (log=-2.192814) P(ddd|Ola)=0.332597 (log=-1.100825) P(ccc|Ola)=0.332597 (log=-1.100825) P(eee|Ola)=0.111602 (log=-2.192814)
2. Wypisz wartości prawdopodobieństw a-priori
P(Ala)=0,571225 (log=-0,559972) P(Ola)=0,428775 (log=-0,846823)
5.2 Predykcja
Sprawdźmy, jakie wartości prawdopodobieństw i etykieta zostaną wyznaczone dla przykładowego wektora cech. (Zakodowane wyrazy to bbb ddd ddd ccc.)
// bbb ddd ccc ddd var testData = new DenseVector(new double[]{1,0,2,1,0});
Dodaj instrukcje:
var proba = model.predictRaw(testData); System.out.println("Pr:["+ Math.exp(proba.apply(0))+", "+Math.exp(proba.apply(1))); var predLabel = model.predict(testData); System.out.println(predLabel);
5.3 Jak są obliczane surowe prawdopodobieństwa
Dla Multininomial NB stosowanym rozkładem jest rozkład nazywany wielomanowym lub wielokrotnym (ang. multinomial)
Jeżeli mamy wektor prawdopodobieństw $[0.2, 0.3,0.1, 0.4]$ to prawdopodobieństwo wystąpienia danych $x=[1,0,2,1]$ wynosi $p(x)=\frac{7!}{1!\cdot 0!\cdot 2! \cdot 4!}\cdot 0.2^1 \cdot 0.3^0 \cdot 0.1^2\cdot 0.4^1$. Przenosimy to także na sytuację, kiedy dane $x$ zostały wyskalowane (nie są liczbami całkowitymi).
Czyli wynikowe prawdopodobieństwo wynosi:
$p(x)=C(x)\prod_{i=1,n}p_i^{x_i}$., gdzie $C(x)$ wyłącznie zależy od $x$ - jest odpowiednikiem $\frac{n!}{x_1!\dots x_n!}$
Logarytmując otrzymujemy:
$log(p(x))=\sum_{i=1,n}x_i\cdot log(p_i) + log(C(x))$
Aby obliczyć $log(p(x))$ wyznaczamy iloczyn skalarny wektora $x$ i wektora $log(p_i)$. Przy porównaniach prawdopodobieństw klas składnik $log(C(x))$ można pominąć, ponieważ, zależy wyłącznie od obserwacji i pojawi się bez zmian w równaniach dla każdej z klas.
1. Oblicz prawdopodobieństwa $p0$, $p1$ odpowiadajace etykietom 0 i 1 zgodnie z powyższym wzorem
System.out.printf(Locale.US,"log(p0)=%g p0=%g log(p1)=%g p1=%g\n", p0,Math.exp(p0), p1,Math.exp(p1)); System.out.println("Wynik klasyfikacj:"+(p0>p1?0:1));
2. Ustaw parametr smoothing klasyfikatora na 0 i sprawdź wyniki
NaiveBayes nb = new NaiveBayes() .setLabelCol("label") .setFeaturesCol("features") .setModelType("multinomial") .setSmoothing(0.0) // <<<<<<<<<<<<< ;
Niestety, obliczenia zaburza P(eee|Ala)=0.000000 (log=-Infinity). Ostatecznie prawdopodobieństwo etykiety 0 ma wartość NaN.
W każdym przypadku, gdy jednym z argumentów porównania jest NaN, zwracana jest wartośc false, stąd rozbieżność wybranych etykiet.
Z tego powodu należy zawsze stosować nawet niewielki parametr wygładzania. W wielu bibliotekach przyjmuje się standardową wartość parametru $alpha=1$. Dla mniejszych wartości są one automatycznie ograniczane od dołu do 1e-10 https://scikit-learn.org/stable/modules/generated/sklearn.naive_bayes.MultinomialNB.html.
6. AuthorRecognitionGridSearchCVNaiveBayes
Wykorzystaj kod klasy dla klasyfikatora DecisionTree. Tworząc siatkę parametrów wybierzemy typ modelu oraz wielkość słownika.
1. Użyj klasyfikatora NaiveBayes
NaiveBayes nb = new NaiveBayes() .setLabelCol("label") .setFeaturesCol("features") .setSmoothing(0.2);
2. Skonfiguruj następująco siatkę parametrów
var scalaIterable = scala.jdk.CollectionConverters. IterableHasAsScala(Arrays.asList("multinomial", "gaussian")).asScala(); ParamMap[] paramGrid = new ParamGridBuilder() .addGrid(countVectorizer.vocabSize(), new int[] {100, 1000,5_000,10_000}) .addGrid(nb.modelType(),scalaIterable ) .build();
3. Uruchom funkcję performGridSearchCV() dla zbioru danych two-books-all-1000-1-stem.csv
4. Odczytaj wartości parametrów
PipelineModel bestModel = (PipelineModel) cvModel.bestModel(); for(var s:bestModel.stages()){ System.out.println(s); }
7. AuthorRecognitionCVNaiveBayes
1. Wykorzystaj wcześniejszy kod dla przeszukiwania siatki parametrów (ale użyj pustej siatki)
2. W funkcji performCV() ustaw wielkość słownika oraz typ klasyfikatora
3. Wykonaj walidację krzyżową i testy dla następujących plików
String filenames[]={ "data/books/two-books-all-1000-1-stem.csv", "data/books/two-books-all-1000-3-stem.csv", "data/books/two-books-all-1000-5-stem.csv", "data/books/two-books-all-1000-10-stem.csv", "data/books/five-books-all-1000-1-stem.csv", "data/books/five-books-all-1000-3-stem.csv", "data/books/five-books-all-1000-5-stem.csv", "data/books/five-books-all-1000-10-stem.csv", };
Podobnie, jak poprzednio podziel plik w proporcji 0.8/0.2 i większą część wykorzystaj w walidacji krzyżowej, a mniejszą do finalnych testów.
4. Zbierz wyniki w postaci tabelki:
- miara F1 dla walidacji krzyżowej na zbiorze treningowym
- accuracy
- precision
- recall
- F1 (dla zbioru testowego)
7. Wnioski
- Który klasyfikator osiąga lepsze wyniki?
- Porównaj czasy uczenia
- Jak efektywność zależy od liczby klas i liczby zdań w dokumentach.